こんにちは!最近勉強し続けているバイオインフォマティクスのアウトプットとして、とりあえずやってみたことを書いていきます。そもそもバイオインフォマティクスって何という方は過去の記事を参照ください。

また、途中謎の単語が多く出てきますがこちらのページにバイオインフォマティクスに関する単語はまとめてあります。ふわっとこんなことができるくらいが分かれば良いと思うので、私から説明はしません。
今回はこのバイオインフォマティクスの中でもメジャーな解析であるトランスクリプトーム解析をやっていきます。これは生体内のmRNAを集めたもの使って遺伝子発現量を解析する手法で例えば創薬分野では次のような効果が期待できます。
- 疾患細胞内の体系的かつ網羅的な遺伝子発現解析
- 医療品開発におけるターゲット候補遺伝子群の同定
- 疾患治療に有意な標的分子の同定
即ち、次のようなことが実現できます。
- 今まで不明だった疾患の原因を解明できる
- 実験コストを大幅に減らすことができ、世の中にいち早く良い薬をだすことができる
上の創薬の例であればヒト(Homo sapiens)が対象になりますが、スケールが膨大でノートパソコンでは耐えられないので普段よく食べているであろうイネ(Oryza satuva)でやってみました。
※実行環境はWindowsのWSLです。
この記事では、データをダウンロードするところまでを書いていきます。
リファレンスゲノムのダウンロード
トランスクリプトーム解析に必要なデータは大きく分けて次の三つです。
- 解析対象の典型的な塩基配列データ(リファレンスゲノム)
- 解析対象となる塩基配列データ(実験データ)
まずはこのリファレンスゲノムをダウンロードします。次世代シーケンサーの発展により、今はありとあらゆる生物種のリファレンスゲノムを無料でダウンロードすることができます。
データベースとして有名なものはUCSC, NCBI, Ensemblがありますが、今回はEnsembl(植物なので Ensembl Plants)からダウンロードします。
まず、ホームページから対象の種を選択します。今回のイネは表示されているのでそこをクリックします。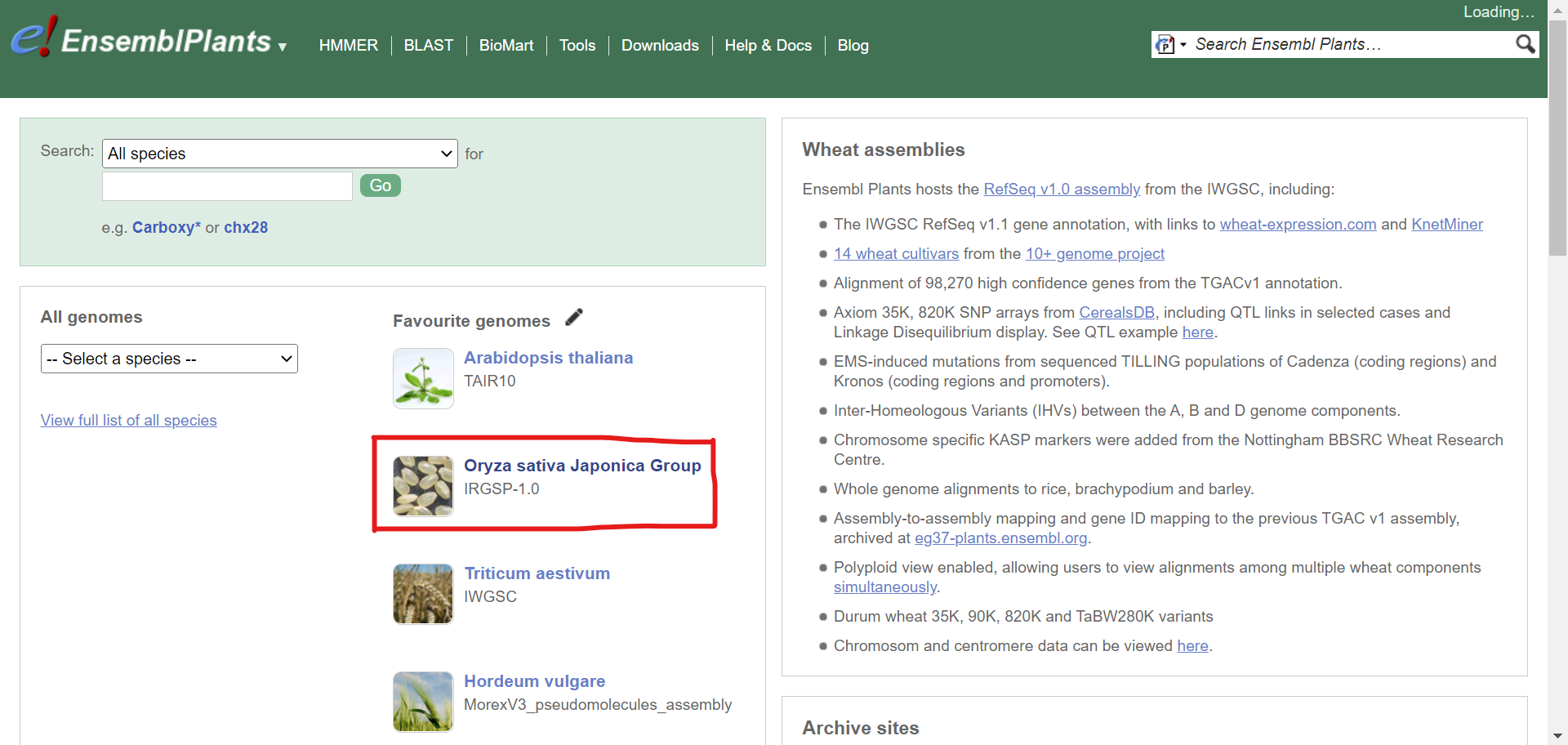
すると、イネのリンクに移動するのでDownloadのリンクをクリックします。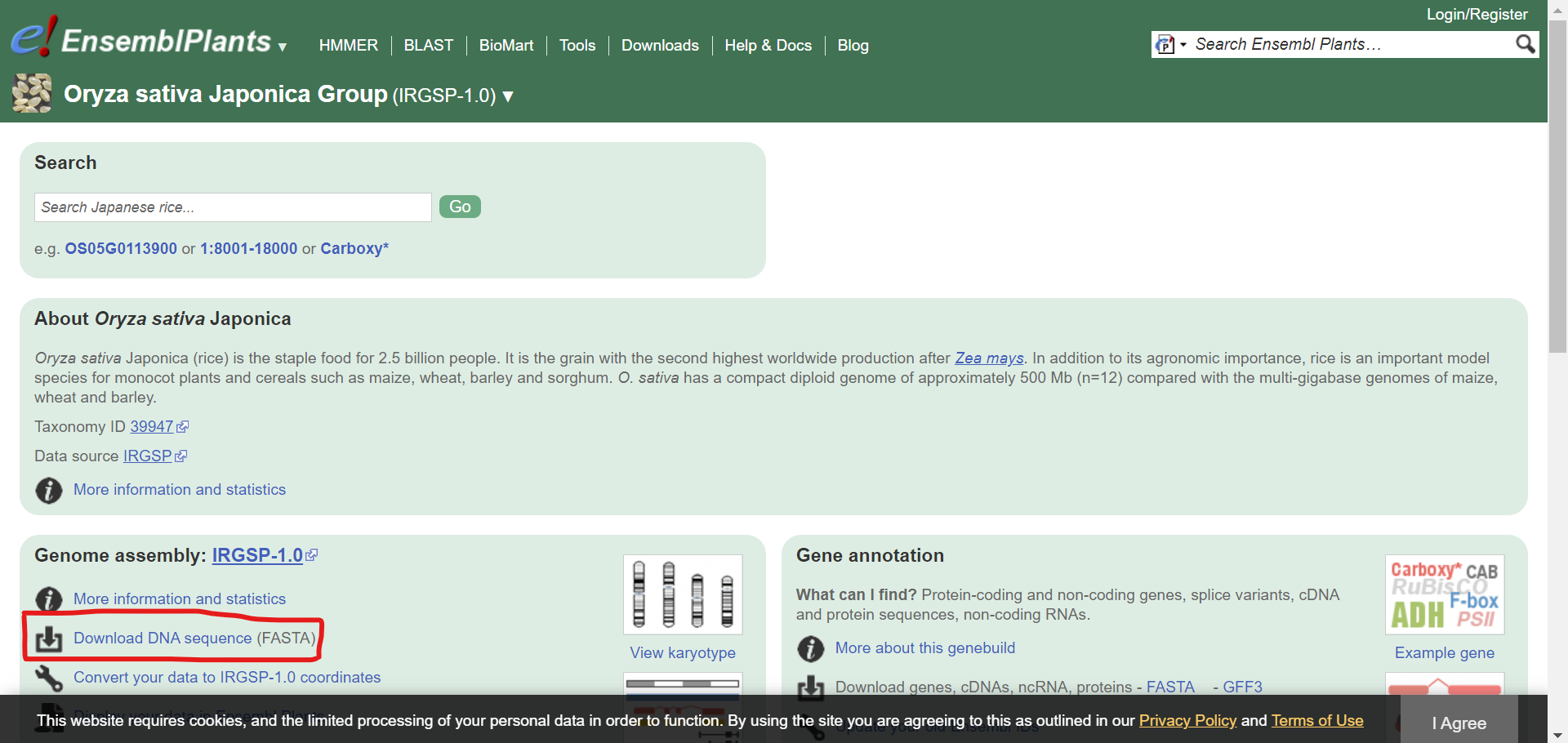
すると、EnsemblのFTPサイトに移動するので、そこのURLリンクをクリックすることでダウンロードすることができます。.faとはFasta形式のファイルであることを表しており塩基配列のデータが格納されています(かなり容量が大きいので圧縮してあります)。染色体別やリピート配列を除去したものなど色んなケースでのゲノム配列が置いてありますが、今回は一番ベーシックな「Oryza_sativa_IRGSP-1.0.dna.toplevel.fa.gz」を選択します。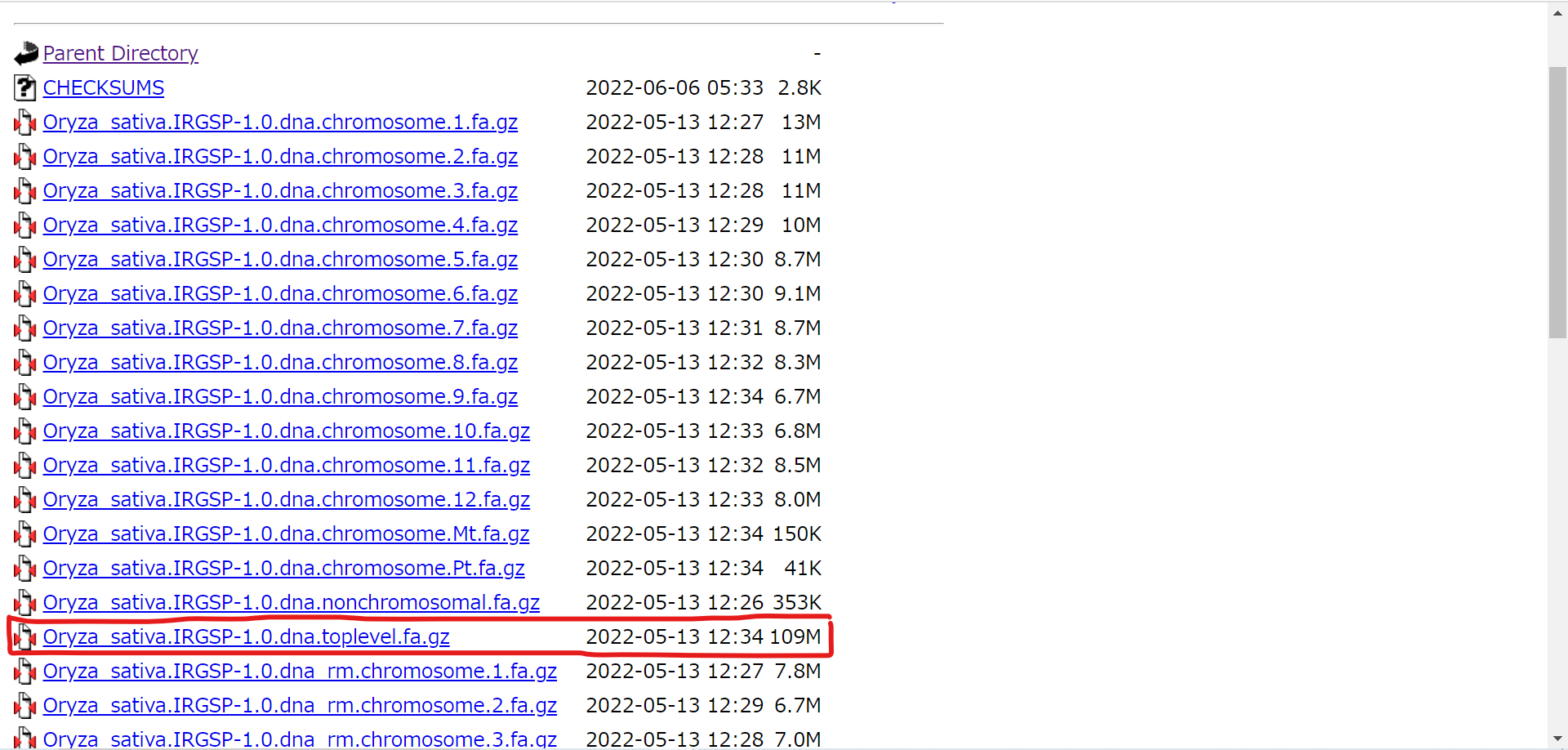 今回はこのゲノム配列に加えて、cDNA配列とアノテーションファイルも同様にダウンロードします。
今回はこのゲノム配列に加えて、cDNA配列とアノテーションファイルも同様にダウンロードします。
※これらはShellコマンドの「wget」などでもインストールできます。
実験データのダウンロード
次に、解析を行う実験データを持ってきます。本来は自分でイネを育ててmRNAを抽出してから次世代シーケンサーで読み取る必要がありますが、普通の分析官にはそれを行う環境はないので Web上で公開されているものから落としてきます。
NCBIデータベースにはこれらのバイオサンプルが大量に格納されていて無料で実験データをダウンロードすることができます。
まずは、一番上の検索でプルダウンメニューを「BioProject」とし、生物種(今回は「Oryza sativa japonica group」)を入力して「Search」をクリックします。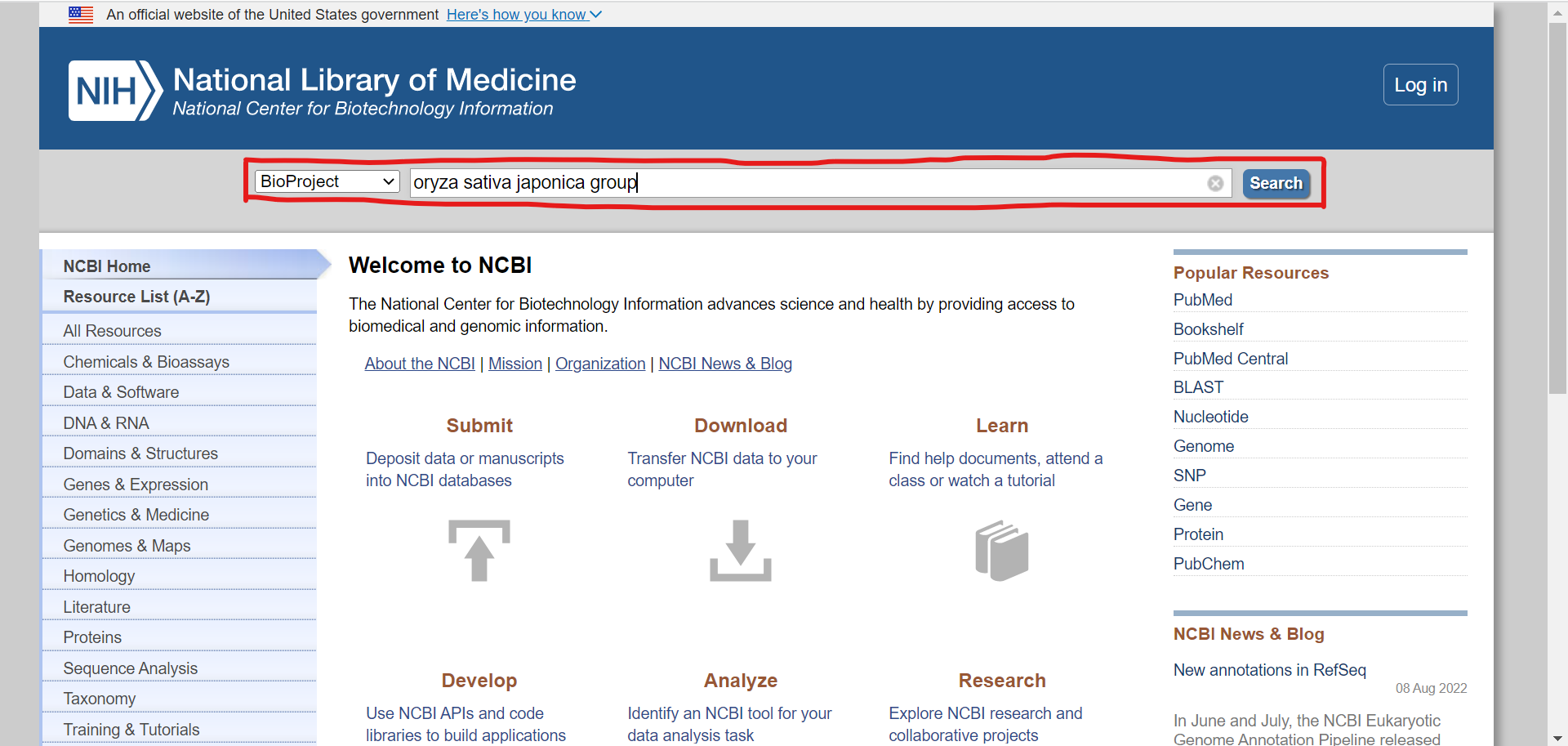
すると、その生物種に対してNCBIの登録があるプロジェクトの一覧が表示されます。ここから気になる実験を選びます。今回は異なる土壌(普通 or 干ばつ地)で栽培された種子のRNAシークエンスデータがあったのでこのデータを使用します。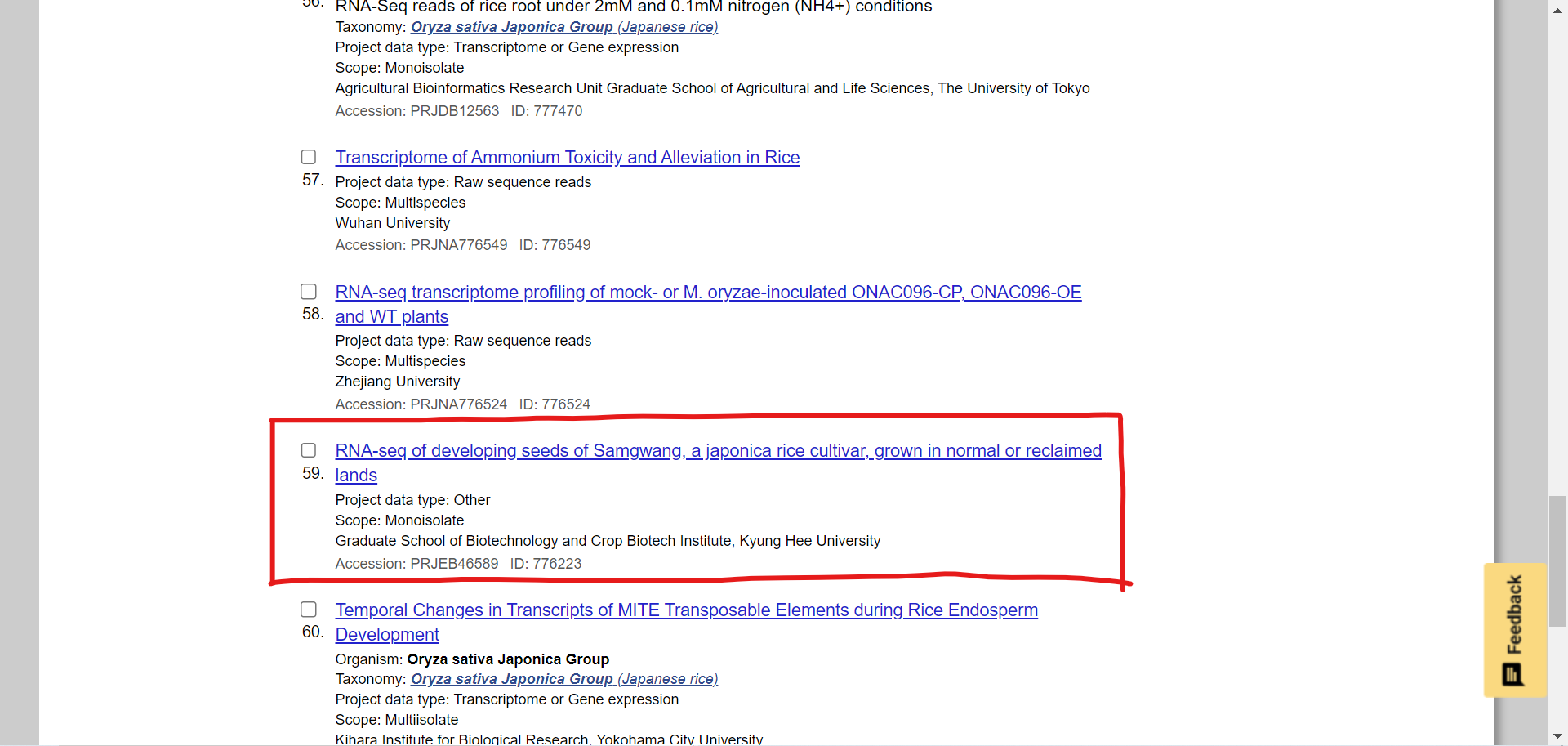
このページを開くと、実験の概要がみられます。RNAシークエンスデータは「SRA Experiments」から探すことができます。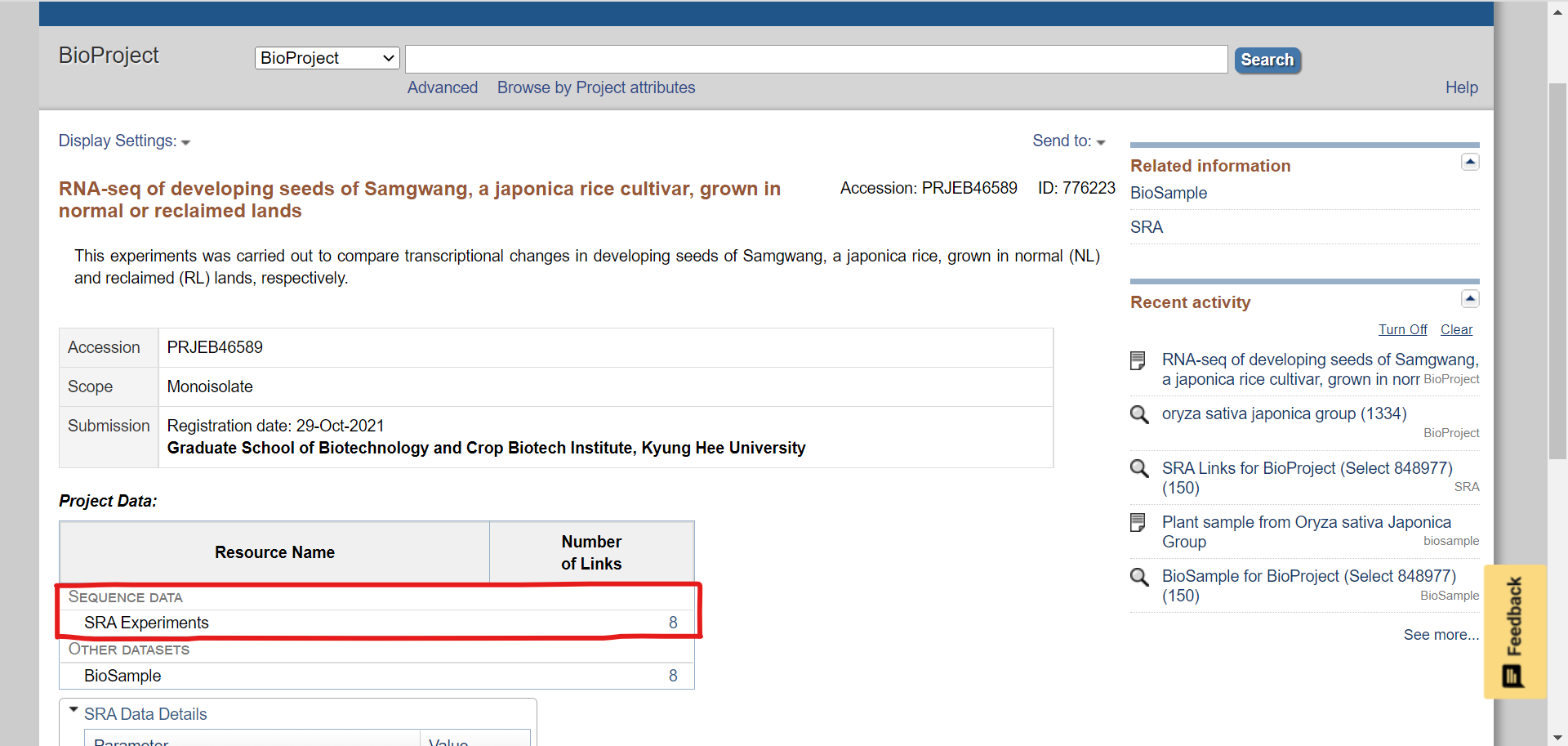
ここで表示されているリンクは全て同じプロジェクト(土壌の実験)のものになるのでどれか一つをクリックします。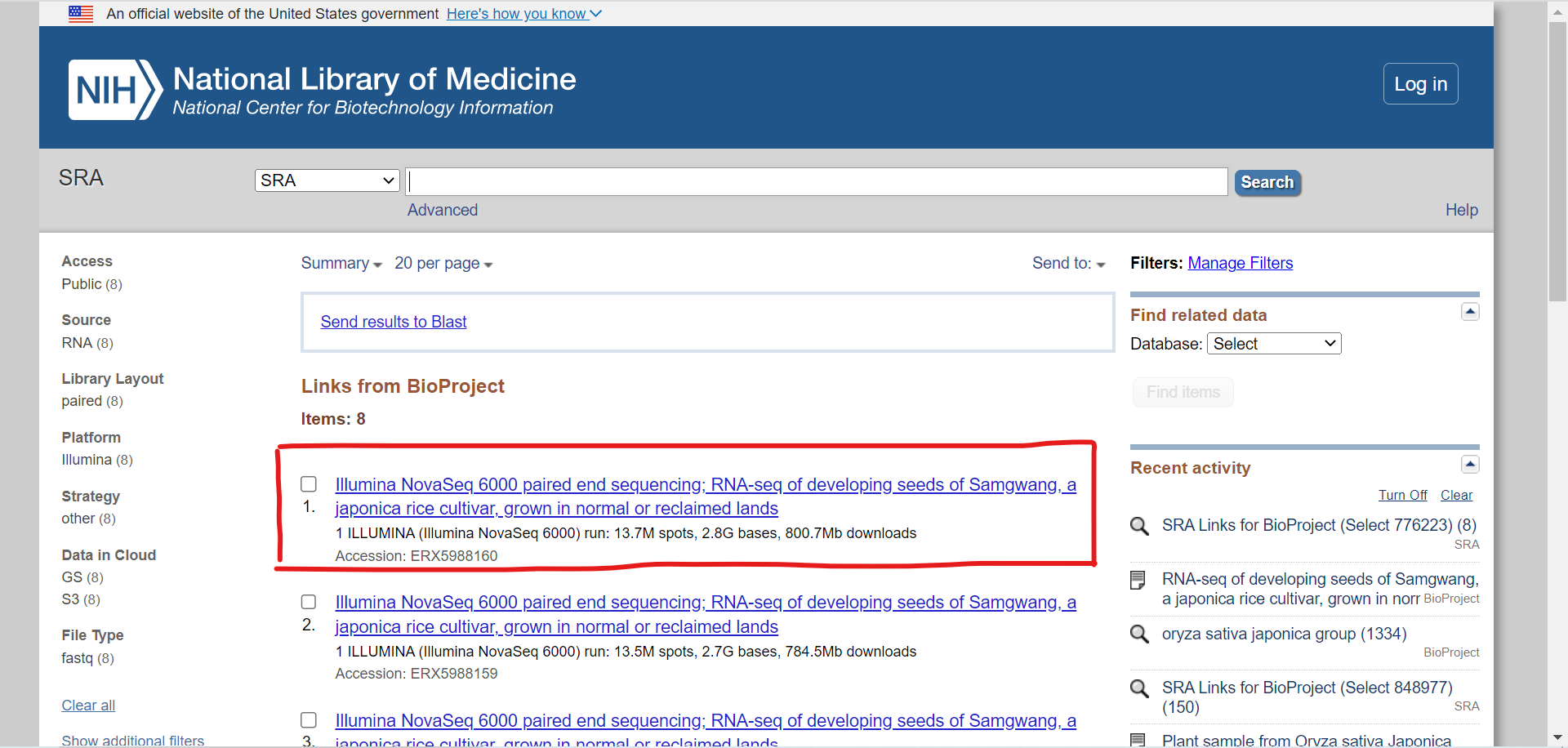
そして、開いたリンクで「Study」の「All runs」をクリックします。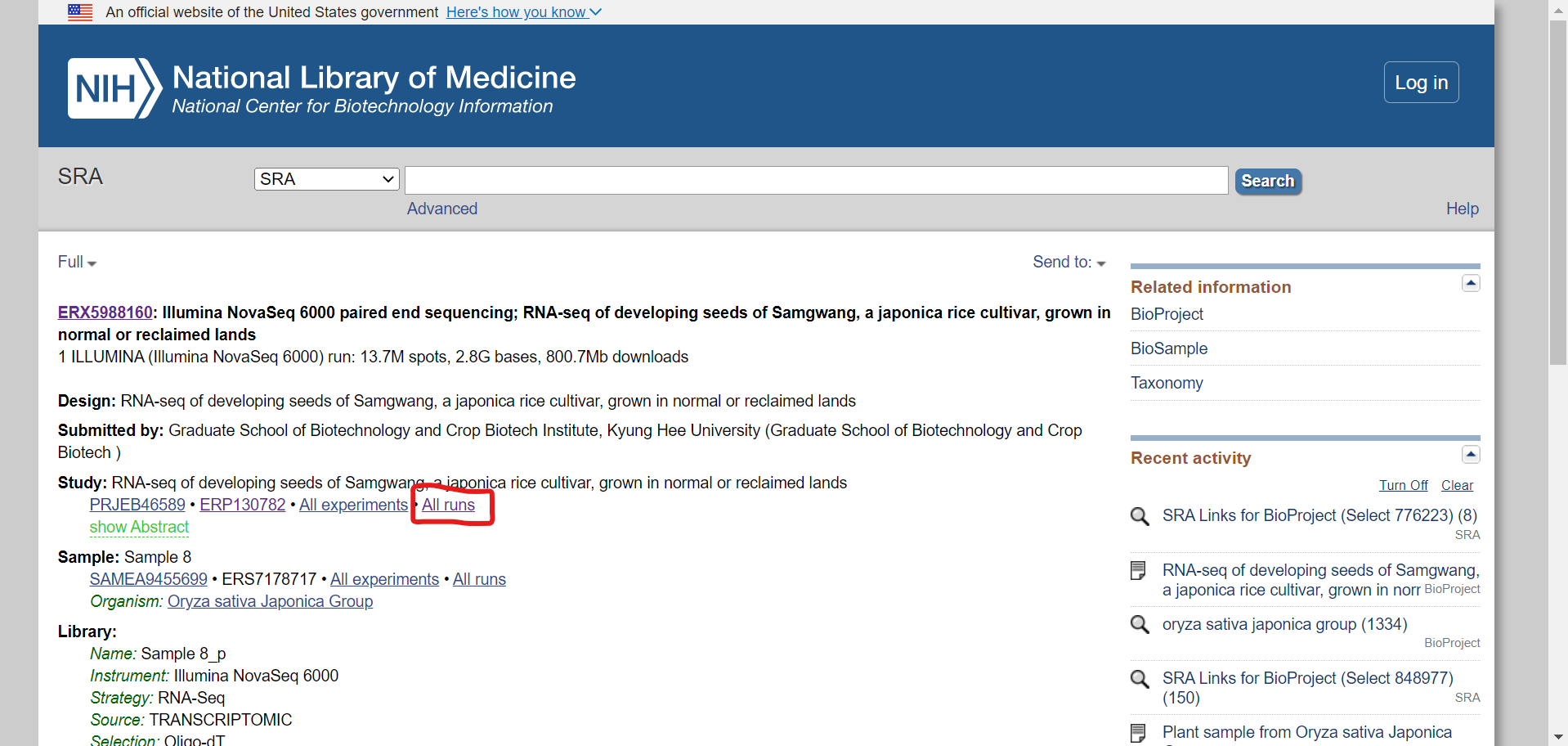
すると実験サンプルのリストを出力できるページに飛びます。ここでは、一つの実験プロジェクトの中でどのサンプルをダウンロードするかを選択することができます。今回は8サンプルなので全てダウンロードします。Totalの「Metadata」と「Accession List」をそれぞれクリックします。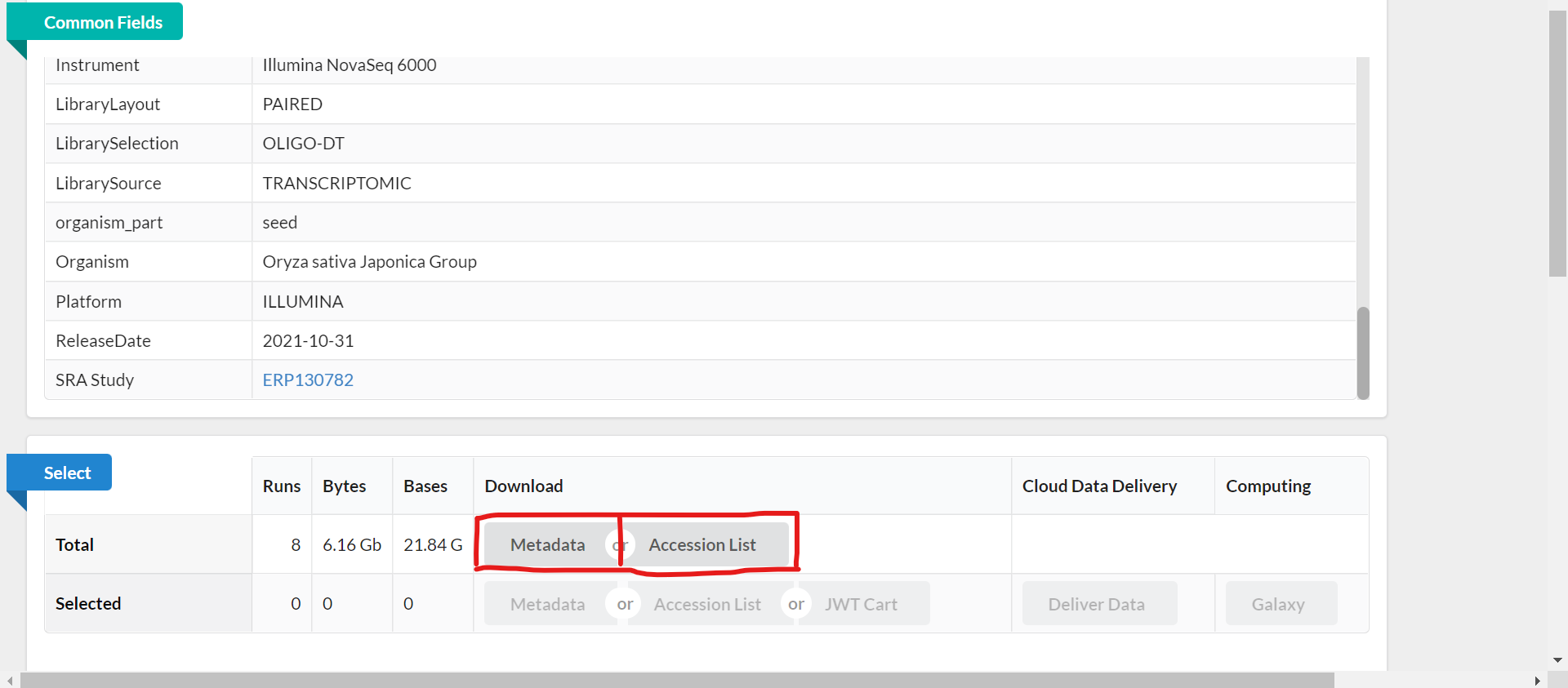
今回でのMetadataとAccession Listをクリックしたときのダウンロードファイルを載せておきます。
Run,Age,Assay Type,AvgSpotLen,Bases,BioProject,BioSample,Broker_name,Bytes,Center Name,common_name,Consent,Cultivar,DATASTORE filetype,DATASTORE provider,DATASTORE region,Developmental_Stage,ENA-FIRST-PUBLIC (run),ENA_first_public,ENA-LAST-UPDATE (run),ENA_last_update,Experiment,External_Id,Genotype,growth_condition,INSDC_center_alias,INSDC_center_name,INSDC_first_public,INSDC_last_update,INSDC_status,Instrument,Library Name,LibraryLayout,LibrarySelection,LibrarySource,organism_part,Organism,Platform,ReleaseDate,Sample Name,sample_name,SRA Study,Submitter_Id
ERR6356602,83 to 90,RNA-Seq,202,2569480804,PRJEB46589,SAMEA9455693,ArrayExpress,783558731,Macrogen,Japanese rice,public,Samgwang,"fastq,sra","gs,s3,ncbi","s3.us-east-1,gs.US,ncbi.public",seed maturation stage,2021-10-28,2021-10-28,2021-10-28,2021-10-28,ERX5988153,SAMEA9455693,wild type genotype,normal land,"Graduate School of Biotechnology and Crop Biotech Institute\, Kyung Hee University","Graduate School of Biotechnology and Crop Biotech Institute\, Kyung Hee University",2021-10-28T13:03:10Z,2021-10-28T13:03:10Z,public,Illumina NovaSeq 6000,Sample 1_p,PAIRED,Oligo-dT,TRANSCRIPTOMIC,seed,Oryza sativa Japonica Group,ILLUMINA,2021-10-31T00:00:00Z,SAMEA9455693,E-MTAB-10774:Sample 1,ERP130782,E-MTAB-10774:Sample 1
ERR6356604,83 to 90,RNA-Seq,202,2737147672,PRJEB46589,SAMEA9455694,ArrayExpress,828768245,Macrogen,Japanese rice,public,Samgwang,"fastq,sra","gs,s3,ncbi","gs.US,ncbi.public,s3.us-east-1",seed maturation stage,2021-10-28,2021-10-28,2021-10-28,2021-10-28,ERX5988155,SAMEA9455694,wild type genotype,normal land,"Graduate School of Biotechnology and Crop Biotech Institute\, Kyung Hee University","Graduate School of Biotechnology and Crop Biotech Institute\, Kyung Hee University",2021-10-28T13:03:10Z,2021-10-28T13:03:10Z,public,Illumina NovaSeq 6000,Sample 3_p,PAIRED,Oligo-dT,TRANSCRIPTOMIC,seed,Oryza sativa Japonica Group,ILLUMINA,2021-10-31T00:00:00Z,SAMEA9455694,E-MTAB-10774:Sample 3,ERP130782,E-MTAB-10774:Sample 3
ERR6356607,83 to 90,RNA-Seq,202,2311782132,PRJEB46589,SAMEA9455697,ArrayExpress,695941088,Macrogen,Japanese rice,public,Samgwang,"sra,fastq","gs,ncbi,s3","ncbi.public,s3.us-east-1,gs.US",seed maturation stage,2021-10-28,2021-10-28,2021-10-28,2021-10-28,ERX5988158,SAMEA9455697,wild type genotype,reclaimed land,"Graduate School of Biotechnology and Crop Biotech Institute\, Kyung Hee University","Graduate School of Biotechnology and Crop Biotech Institute\, Kyung Hee University",2021-10-28T13:03:10Z,2021-10-28T13:03:10Z,public,Illumina NovaSeq 6000,Sample 6_p,PAIRED,Oligo-dT,TRANSCRIPTOMIC,seed,Oryza sativa Japonica Group,ILLUMINA,2021-10-31T00:00:00Z,SAMEA9455697,E-MTAB-10774:Sample 6,ERP130782,E-MTAB-10774:Sample 6
ERR6356603,83 to 90,RNA-Seq,202,3280831278,PRJEB46589,SAMEA9455692,ArrayExpress,994920315,Macrogen,Japanese rice,public,Samgwang,"sra,fastq","ncbi,s3,gs","s3.us-east-1,gs.US,ncbi.public",seed maturation stage,2021-10-28,2021-10-28,2021-10-28,2021-10-28,ERX5988154,SAMEA9455692,wild type genotype,normal land,"Graduate School of Biotechnology and Crop Biotech Institute\, Kyung Hee University","Graduate School of Biotechnology and Crop Biotech Institute\, Kyung Hee University",2021-10-28T13:03:10Z,2021-10-28T13:03:10Z,public,Illumina NovaSeq 6000,Sample 2_p,PAIRED,Oligo-dT,TRANSCRIPTOMIC,seed,Oryza sativa Japonica Group,ILLUMINA,2021-10-31T00:00:00Z,SAMEA9455692,E-MTAB-10774:Sample 2,ERP130782,E-MTAB-10774:Sample 2
ERR6356605,83 to 90,RNA-Seq,202,2959762378,PRJEB46589,SAMEA9455695,ArrayExpress,898142235,Macrogen,Japanese rice,public,Samgwang,"fastq,sra","s3,gs,ncbi","s3.us-east-1,ncbi.public,gs.US",seed maturation stage,2021-10-28,2021-10-28,2021-10-28,2021-10-28,ERX5988156,SAMEA9455695,wild type genotype,normal land,"Graduate School of Biotechnology and Crop Biotech Institute\, Kyung Hee University","Graduate School of Biotechnology and Crop Biotech Institute\, Kyung Hee University",2021-10-28T13:03:10Z,2021-10-28T13:03:10Z,public,Illumina NovaSeq 6000,Sample 4_p,PAIRED,Oligo-dT,TRANSCRIPTOMIC,seed,Oryza sativa Japonica Group,ILLUMINA,2021-10-31T00:00:00Z,SAMEA9455695,E-MTAB-10774:Sample 4,ERP130782,E-MTAB-10774:Sample 4
ERR6356606,83 to 90,RNA-Seq,202,2472410512,PRJEB46589,SAMEA9455696,ArrayExpress,751175813,Macrogen,Japanese rice,public,Samgwang,"fastq,sra","s3,ncbi,gs","s3.us-east-1,gs.US,ncbi.public",seed maturation stage,2021-10-28,2021-10-28,2021-10-28,2021-10-28,ERX5988157,SAMEA9455696,wild type genotype,reclaimed land,"Graduate School of Biotechnology and Crop Biotech Institute\, Kyung Hee University","Graduate School of Biotechnology and Crop Biotech Institute\, Kyung Hee University",2021-10-28T13:03:10Z,2021-10-28T13:03:10Z,public,Illumina NovaSeq 6000,Sample 5_p,PAIRED,Oligo-dT,TRANSCRIPTOMIC,seed,Oryza sativa Japonica Group,ILLUMINA,2021-10-31T00:00:00Z,SAMEA9455696,E-MTAB-10774:Sample 5,ERP130782,E-MTAB-10774:Sample 5
ERR6356608,83 to 90,RNA-Seq,202,2729312698,PRJEB46589,SAMEA9455698,ArrayExpress,822638637,Macrogen,Japanese rice,public,Samgwang,"sra,fastq","ncbi,s3,gs","s3.us-east-1,gs.US,ncbi.public",seed maturation stage,2021-10-28,2021-10-28,2021-10-28,2021-10-28,ERX5988159,SAMEA9455698,wild type genotype,reclaimed land,"Graduate School of Biotechnology and Crop Biotech Institute\, Kyung Hee University","Graduate School of Biotechnology and Crop Biotech Institute\, Kyung Hee University",2021-10-28T13:03:10Z,2021-10-28T13:03:10Z,public,Illumina NovaSeq 6000,Sample 7_p,PAIRED,Oligo-dT,TRANSCRIPTOMIC,seed,Oryza sativa Japonica Group,ILLUMINA,2021-10-31T00:00:00Z,SAMEA9455698,E-MTAB-10774:Sample 7,ERP130782,E-MTAB-10774:Sample 7
ERR6356609,83 to 90,RNA-Seq,202,2777289718,PRJEB46589,SAMEA9455699,ArrayExpress,839639001,Macrogen,Japanese rice,public,Samgwang,"sra,fastq","ncbi,s3,gs","s3.us-east-1,gs.US,ncbi.public",seed maturation stage,2021-10-28,2021-10-28,2021-10-28,2021-10-28,ERX5988160,SAMEA9455699,wild type genotype,reclaimed land,"Graduate School of Biotechnology and Crop Biotech Institute\, Kyung Hee University","Graduate School of Biotechnology and Crop Biotech Institute\, Kyung Hee University",2021-10-28T13:03:10Z,2021-10-28T13:03:10Z,public,Illumina NovaSeq 6000,Sample 8_p,PAIRED,Oligo-dT,TRANSCRIPTOMIC,seed,Oryza sativa Japonica Group,ILLUMINA,2021-10-31T00:00:00Z,SAMEA9455699,E-MTAB-10774:Sample 8,ERP130782,E-MTAB-10774:Sample 8Metadata > SraRunTable.txt(growth_conditionが土壌についてのカラム)
ERR6356602
ERR6356604
ERR6356607
ERR6356603
ERR6356605
ERR6356606
ERR6356608
ERR6356609Accession List > SRR_Acc_List.txt
ここからはShellコマンドでRNAシークエンスデータをダウンロードしていきます。ここではWebから直接ダウンロードする方法がなく、SRA-toolkitを使う必要があります。
ダウンロードしたら、先ほどダウンロードしたSRR_Acc_List.txtを利用してRNAシークエンスデータをダウンロードします。
詳しい手順はこちらを参照してください。
ここまででトランスクリプトーム解析に必要なデータをダウンロードしました。次回以降で実際に解析をしていきたいと思います。
GRIでは今回扱ったバイオデータに関わらず様々なデータに関する支援が可能です。何かお困りごとや一緒に仕事をしてみたいという方は弊社コーポレートサイトからご連絡ください
最後まで読んでいただきありがとうございました。















